PS:这个数据库需要科学上网才能用。
使用方法
使用方法很简单,网站提供了三种检索方式:基本检索; 高级检索以及列表批量检索。三种检索方式大同小异,这里就介绍一下基本检索。
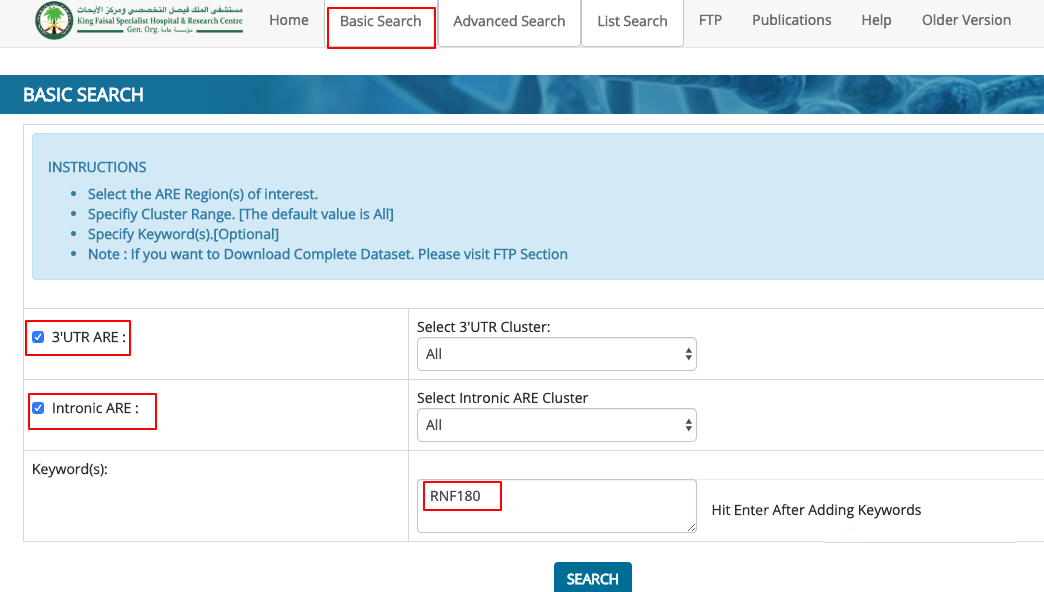
首先需要我们去选择的是,想看3UTR区还是内含子区的ARE。我们把两个都选上。
另外直接输入检索的关键词即可。最基本的还是基因名,我们这里输入:
RNF180。点击搜索即可。
>>>> 结果解读
检索完之后,我们可以得到关于这个基因是否有ARE的信息。如下图:
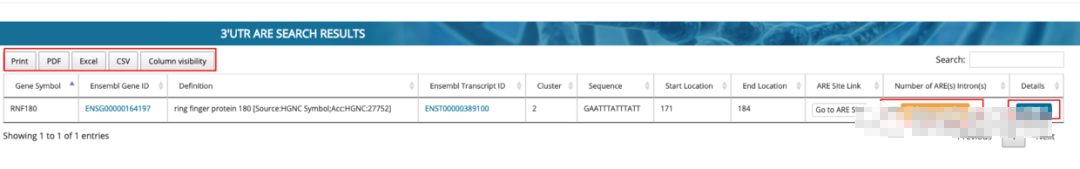
结果中如果3’UTR区和内含子区都有调控位点。默认显示的是3’UTR的结果。我们可以通过切换成内含子的结果。对于结果的检索网站还同时提供了结果的下载。
另外,这个网站目前就提供了最基本的查询信息。如果还要查看进一步的信息。需要去他们开发的另外一个网站:AREsite2(http://rna.tbi.univie.ac.at/AREsite2/welcome)
AREsite2

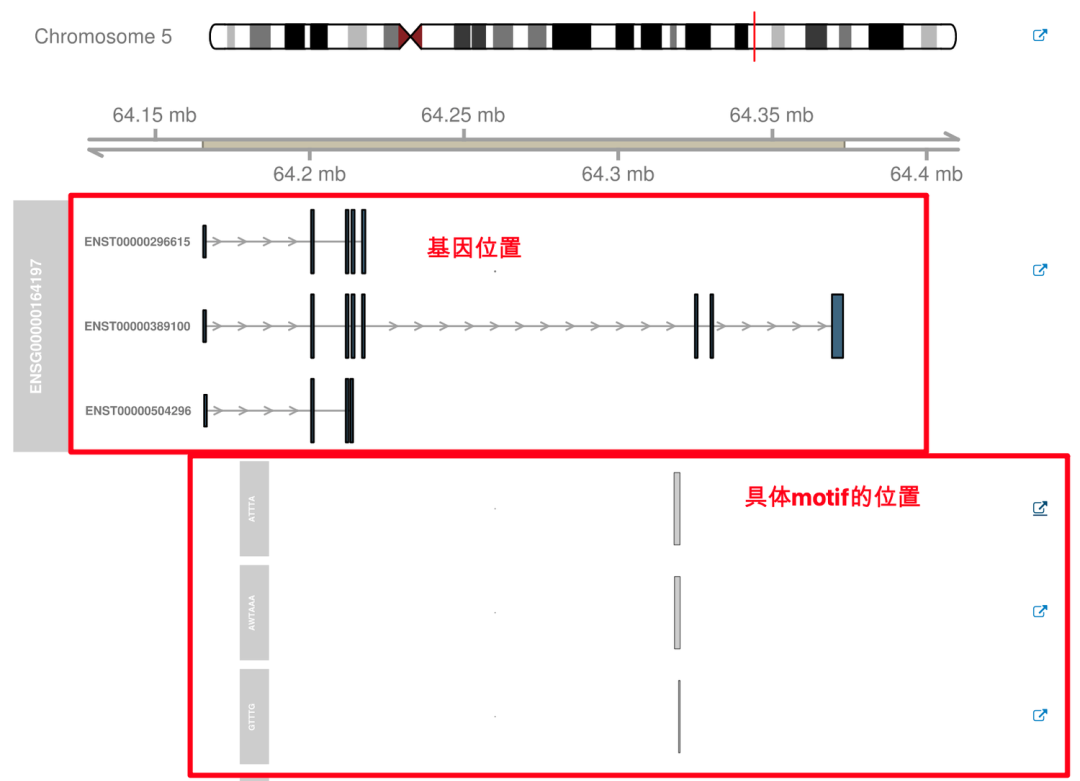
该网站的使用也是很简单的。我们需要做的就是:
选择感兴趣的motif
选择物种
选择感兴趣的区域
输入想要查看的基因名:我们这里还是选择RNF180


数据下载
关于ARED-Plus数据库,如果觉得每次上网站检索很麻烦的话,网站提供了把所有数据下载下来的功能。我们选择 FTP即可看到下载所有数据的界面。
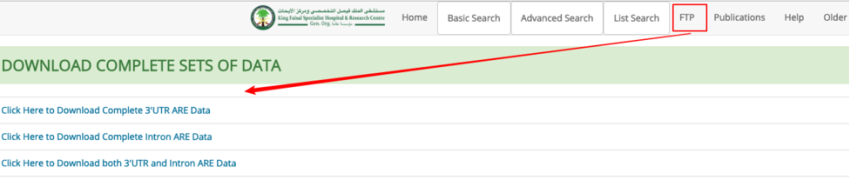
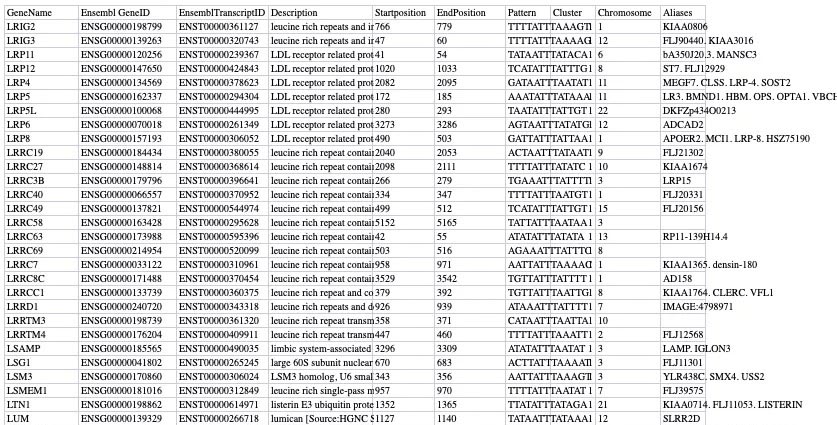
下载下来的数据是这个样子的,信息还是挺全的。这样假如我有整个基因组很多靶向基因进行分析的时候,就可以批量进行个性化检索或者处理了。